Southern Blot

Beim Southern Blot, auch Southern-Blot-Hybridisierungsverfahren genannt, handelt es sich um eine 1975 von Edwin M. Southern entwickelte molekularbiologische Untersuchungsmethode für die DNA.[1] Sie ermöglicht den Nachweis einer Gensequenz in einem komplexen DNA-Gemisch (z. B. dem gesamten Genom eines Organismus) innerhalb kurzer Zeit, ohne dass sämtliche Sequenzen des Gemisches entschlüsselt werden müssen.
Funktionsweise
[Bearbeiten | Quelltext bearbeiten]Die zu untersuchende DNA wird mit einem oder mehreren Restriktionsenzymen behandelt und anschließend durch Gelelektrophorese der Größe nach aufgetrennt. Die DNA-Fragmente werden durch Alkalien in Einzelstränge gespalten und das im Gel entstandene Trennmuster auf eine Membran (meist Nylon oder Nitrocellulose) übertragen (Blotten) und dort dauerhaft fixiert.
Anschließend wird die Membran mit einer chemisch oder radioaktiv markierten Gensonde behandelt. Diese Sonde besteht aus einzelsträngiger RNA (RNA-DNA-Hybride sind stabiler als DNA-DNA-Hybride), welche zur gesuchten Sequenz komplementär ist oder aus doppelsträngiger DNA, die vor der Hybridisierung durch Erhitzen denaturiert wird. Befindet sich diese Sequenz irgendwo auf der Membran, so bildet die Sonde Basenpaarungen mit dieser aus und bindet dauerhaft in diesem Bereich (Hybridisierungsvorgang).
Alle unspezifischen Bindungen werden anschließend abgewaschen. Je nach Markierung der Sonde erfolgt die Detektion z. B. durch Auflegen eines Röntgenfilms und von Verstärkerfolien oder von Röntgenspeicherfolien (als Autoradiographie bei radioaktiven Markierungen) in einer lichtgeschützten Kassette. Ist die Sonde an ein Enzym gekoppelt, so kann die enzymatische Reaktion auf der Membran detektiert werden.
Versuchsablauf
[Bearbeiten | Quelltext bearbeiten]Vorbehandlung der DNA
[Bearbeiten | Quelltext bearbeiten]Bei der zu untersuchenden DNA handelt es sich meist um genomische DNA. Sie wird aus den Zellen isoliert und mit einem oder mehreren Restriktionsenzymen behandelt. Je nachdem, wie oft die gewählten Enzyme in der gesuchten Sequenz schneiden, entstehen unterschiedlich viele detektierbare Fragmente. Liegt zum Beispiel keine Schnittstelle in der Sequenz, so entsteht eine Bande, schneiden die Enzyme einmal entstehen zwei Banden. (Ausnahmen bilden hier natürlich Plasmide, bei denen sich zwei Banden erst bei zwei Schnittstellen ergeben, da sie zunächst als Ring vorliegen.)
Das gilt, wenn sich die gesuchte Sequenz nur an einer Stelle in der genomischen DNA befindet oder sich die Sequenzen, wie es im Falle eines diploiden Organismus fast immer der Fall ist, nicht in ihrer Fragmentlänge unterscheiden. Bei einer quantitativen Analyse erlaubt die Signalstärke Hinweise auf die Kopienzahl. Daher sollten transgene Organismen durch Southern-Blot untersucht werden, da die Zahl der Signale Hinweise auf die Zahl der Integrationsorte und die Intensität Rückschlüsse auf die Zahl der Integrate erlaubt.

Gelelektrophorese
[Bearbeiten | Quelltext bearbeiten]Nach dieser Vorbehandlung trennt man zwischen 5 µg und 30 µg der DNA zusammen mit einem Marker in einem Agarosegel der Größe nach auf. Da die Restriktionsenzyme statistisch verteilt in der DNA schneiden, gibt es Fragmente jeder Größe. In dem Gel entstehen so keine Banden, sondern eine gleichmäßige Verteilung der DNA, ein Schmier.
Nach der Elektrophorese wird ein fluoreszierendes Lineal neben den Marker auf das zuvor mit Ethidiumbromid angefärbte Gel gelegt und unter UV-Licht fotografiert. Man weiß nun, welche Fragmente auf Grund ihrer Größe wie weit im Gel gelaufen sind. Das ist notwendig, da der Marker nach der Detektion mit der Sonde nicht mehr sichtbar ist. Man kann später durch einfaches Ausmessen mit einem Lineal die Molekülmasse der sichtbar gewordenen Fragmente bestimmen.
Das Gel wird anschließend nacheinander mit stark verdünnter Salzsäure, einer Denaturierungslösung und einer Neutralisierungslösung behandelt, um es auf den DNA-Transfer vorzubereiten.
Blotting
[Bearbeiten | Quelltext bearbeiten]Als Blotting wird der Transfer der DNA aus dem Gel auf eine Membran bezeichnet. Dafür gibt es verschiedene Möglichkeiten:
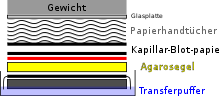
- Kapillar-Blot
- Die treibende Kraft ist ein Flüssigkeitsstrom, der von einem Reservoir ausgehend von unten durch das Gel, weiter durch die Membran zu einem Stapel saugfähigen Materials läuft. Die Flüssigkeit ist meistens eine alkalische Pufferlösung, welche die DNA wieder in Einzelstränge zerlegt. Dieser Strom zieht die DNA aus dem Gel mit, die anschließend in den Maschen der Membran hängen bleibt. Das Verfahren läuft meist 10 bis 12 Stunden (über Nacht). In dieser Zeit können durch ein Gel von 15 mal 15 Zentimeter bis zu 2 Liter Salzlösung laufen. Wichtig ist, dass sich nirgendwo im Aufbau Luftblasen befinden, da sie den Flüssigkeitsstrom unterbrechen und an dieser Stelle die DNA nicht übertragen wird.
- Vakuum-Blot
- Er funktioniert prinzipiell wie der Kapillar-Blot. Statt des saugfähigen Materials zieht hier allerdings ein Unterdruck die Flüssigkeit durch Gel und Membran. Der Vakuum-Blot ist schneller und sparsamer, was die Salzlösung angeht.
- Elektro-Blot
- Beim Elektroblot wird die negative Ladung der DNA genutzt. Das Gel liegt auf einer Kathodenplatte. Auf dem Gel liegt die Membran und darüber die Anodenplatte. Eine Salzlösung gewährleistet, dass ein elektrischer Strom fließen kann und die DNA sich in Richtung der Anode bewegt. Sie wandert aus dem Gel und bleibt auf der Membran hängen.
Nach dem Blotting schwenkt man die Membran in niedrig konzentriertem „Salzpuffer“. Anschließend kann durch Vernetzung mit UV-Licht die DNA in der Membran dauerhaft fixiert werden – das crosslinking. Will man nicht gleich mit der Analyse fortfahren, wird sie in Folie eingeschweißt und im Kühlschrank gelagert oder zwischen Blotting-Filterpapier bei Raumtemperatur gelagert. Man kann auch an Stelle des crosslinkings die Membran 2–3 Stunden bei 80 °C „backen“.
Blockierung
[Bearbeiten | Quelltext bearbeiten]Die nach dem Transfer verbliebenen freien DNA-Bindungsstellen auf der Membran werden meistens mit einer Blockierungslösung mit hitzedenaturierter DNA in zehnfach konzentrierter Denhardt-Lösung mit dem Tensid SDS (1 % m/V) in einem Phosphat-Puffer inkubiert (zu 50 Mikrogramm pro Milliliter, für fünf Stunden bei 42 °C), wodurch die DNA-bindenden Stellen auf der Membran abgesättigt werden. Dazu wird meistens eine vergleichsweise kostengünstige DNA aus Lachs- oder Heringssperma verwendet. Das Tensid mindert unspezifische Bindungen von Nicht-DNA-Molekülen an die Membran. Durch die Blockierung (synonym Prähybridisierung) werden unspezifische Bindungen der Sonde über die gesamte Membran vermieden, die sonst zu einer starken Hintergrundfärbung führen würde.
Synthese der Sonde
[Bearbeiten | Quelltext bearbeiten]Die Synthese der DNA-Sonde geschieht enzymatisch entweder durch PCR, Random Priming oder Nick translation. Man legt die Zielsequenz vor und kopiert sie sehr häufig (PCR), setzt sie als Matrize (Template) ein (Random Priming) oder modifiziert sie (Nick translation). Dabei werden die später zur Detektion genutzten Bausteine eingebaut. Im Falle einer RNA-Sonde erfolgt die Synthese mittels in vitro-Transkription mit Bakteriophagen RNA-Polymerasen, wie der T7-RNA-Polymerase. Die zwei gängigsten Markierungen sind:
- α-32P-dATP-Markierung
- Die DNA wird aus den vier Grundbausteinen dATP, dTTP, dGTP und dCTP aufgebaut. Das dATP trägt für diesen Versuch das radioaktive Phosphorisotop 32P. Bei der Synthese wird es in die Sonde eingebaut, die dadurch ständig Betastrahlen aussendet. Analog wird bei der RNA-Synthese ATP, GTP, CTP und α-32P-UTP eingesetzt. Legt man die Membran auf einen Röntgenfilm, wird dieser an den Stellen mit der Sonde geschwärzt. Eine moderne digitale Alternative ist das Auflegen auf eine Röntgenspeicherfolie. Dabei handelt es sich um eine spezielle Platte, deren Leuchtstoffschicht durch Strahlung nachhaltig angeregt wird. Diese Veränderung kann mit einem Lesegerät, einem Phosphorimager (auch Phosphoimager), durch Anregung mit Laserlicht in den Computer eingelesen und dort weiter bearbeitet werden. Die Platte kann anschließend regeneriert und erneut verwendet werden
- DIG-Markierung
- In die DNA wird während des Kopierens an manchen Stellen dUTP statt dTTP eingebaut. An diesem Baustein hängt das Glycosid Digoxigenin. Analog wird bei der in vitro-Transkription DIG-UTP eingesetzt. Man kann nun dieses Digoxigenin mit einem spezifischen anti-DIG-Antikörper, an dem ein Enzym gekoppelt ist, binden. Dieses katalysiert eine Licht- oder Farb-Reaktion. Die Detektion der Chemolumineszenz erfolgt durch Auflegen der Membran auf Fotopapier. Die Stellen, an denen die Sonde liegt, werden durch das entstehende Licht geschwärzt. Diese Methode hat den Vorteil, dass nicht mit radioaktivem Material gearbeitet wird.
Parallel zur DIG-Markierung gibt es noch andere chemische Markierungssysteme, wie etwa das Biotin/Streptavidin-System, bei dem ein sichtbarer Farbstoff gebildet wird. Allen chemischen Markern ist gemein, dass sie im Vergleich zur radioaktiven Markierung weniger sensitiv sind. Trotzdem finden sie im Laboralltag Anwendung, wenn etwa kein Isotopenlabor zur Verfügung steht.
Hybridisierung
[Bearbeiten | Quelltext bearbeiten]Die Sonde, in der Regel 50 µl DNA-Lösung, wird direkt vor der Verwendung auf 94 °C erhitzt. Die Hitze führt zur Trennung der doppelsträngigen DNA. Nur so kann sie mit der DNA auf der Membran Basenpaarungen eingehen. Die Sondenlösung wird dann sofort mit 5 bis 10 ml einer Hybridisierungslösung gemischt und zusammen mit der Membran in eine Glasröhre gegeben. In einem Ofen wird diese Röhre bei 40 bis 60 °C für mehrere Stunden automatisch gewendet. Die hohe Temperatur garantiert, dass sich die Sonde nur an die gesuchte Zielsequenz bindet und keine unspezifischen Wechselwirkungen mit anderen Sequenzen oder Membranteilen eingeht.
Die genaue zu wählende Temperatur ist allerdings abhängig von der Länge und dem G/C-Gehalt der Sonde; außerdem von der Salzkonzentration der Lösung. Eine Formel zum Berechnen lautet wie folgt: Tm = 81,5 °C + 0,41 × (%G/C) + 16,6 log [Na+] − 500/n − 0,61 * (%Formamid); wobei n = Anzahl der Sondenbasen; Formamid ist ein häufiger Bestandteil von Hybridisierungslösungen.[2]
Anschließend wird die Membran mehrmals mit Salzlösungen niedriger Konzentration und bei Temperaturen bis 65 °C gewaschen, um den ungebundenen Anteil der Sonde restlos zu entfernen. Es folgt die Detektion je nach Markierung.
Stripping
[Bearbeiten | Quelltext bearbeiten]Stripping nennt man das Entfernen der spezifisch gebundenen Sonde. Nach der Detektion ist es möglich, die Membran in eine Stripping-Lösung zu tauchen und mehrere Minuten auf 94 °C zu erhitzen. Die Basenpaarungen zwischen Sonde und Zielsequenz werden durch die Hitze aufgebrochen und die Sonde löst sich ab. Die Membran kann jetzt erneut vorhybridisiert und anschließend mit einer anderen Sonde behandelt werden. Dieser Kreislauf kann je nach der Menge der übertragenen DNA auf der Membran, der Qualität der Membran und der Lösungen bis zu 20 Mal wiederholt werden.
Ähnliche Methoden
[Bearbeiten | Quelltext bearbeiten]Analog zum Southern Blot gibt es auch einen Northern Blot. Hier wird RNA durch Gelelektrophorese aufgetrennt und auf eine Membran übertragen. Mit dem Northern-Blot kann der Expressionsstatus eines Gens überprüft werden.
Beim Western Blot werden Proteine durch eine etwas andere Elektrophorese (PAGE – Polyacrylamid-Gel-Elektrophorese) aufgetrennt und auf die Membran übertragen. Als Sonde dienen hier spezifische Antikörper. Hier kann man jedoch auch Protein-Interaktionen untersuchen, wobei dann der jeweilige Ligand per Antikörper detektiert wird (Far-Western-Blotting). Die Kombination aus Western und Southern Blot wird als Southwestern Blot bezeichnet.
Literatur
[Bearbeiten | Quelltext bearbeiten]Weitere Literatur
[Bearbeiten | Quelltext bearbeiten]- Thomas Maniatis, Edward F. Fritsch, Joseph Sambrook: Molecular cloning. A laboratory manual. Cold Spring Harbor Laboratory, Cold Spring Harbour NY 1982, ISBN 0-87969-136-0.
Quellenangaben
[Bearbeiten | Quelltext bearbeiten]- ↑ E. M. Southern: Detection of specific sequences among DNA fragments separated by gel electrophoresis. In: Journal of Molecular Biology. Bd. 98, Nr. 3, 1975, S. 503–517, PMID 1195397, doi:10.1016/S0022-2836(75)80083-0.
- ↑ Judy Meinkoth, Geoffrey Wahl: Hybridization of nucleic acids immobilized on solid supports. In: Analytical Biochemistry. Bd. 138, Nr. 2, May 1984, S. 267–284, doi:10.1016/0003-2697(84)90808-X.
Siehe auch
[Bearbeiten | Quelltext bearbeiten]Weblinks
[Bearbeiten | Quelltext bearbeiten]- Southern Blot Animation (engl.; SWF-Datei; 511 kB)
- detaillierte Durchführung eines Southern Blots ( vom 30. April 2008 im Internet Archive) (engl.)
- Schema eines Southern Blots ( vom 10. April 2008 im Internet Archive)